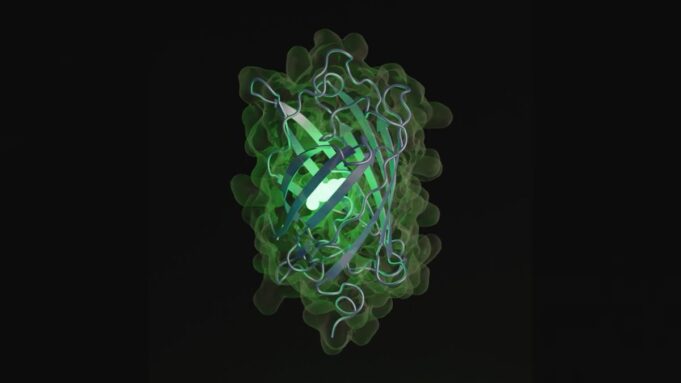
Un inteligencia artificial (AI) El modelo ha simulado medio billón de años de evolución molecular para crear el código para una proteína previamente desconocida, según un nuevo estudio. La proteína brillante, que es similar a las que se encuentran en las medusas y los corales, puede ayudar en el desarrollo de nuevos medicamentos, dicen los investigadores.
Las proteínas son uno de los componentes básicos de la vida y realizan diversas funciones en el cuerpo, como construir músculos y lucha contra la enfermedad. La proteína simulada, llamada ESMGFP, solo existe como código de computadora, pero contiene el plan para un tipo previamente desconocido de proteína fluorescente verde. En la naturaleza, las proteínas fluorescentes verdes le dan a las medusas fluorescentes y los corales su brillo.
La secuencia de letras que explican las instrucciones para hacer ESMGFP es solo un 58% similar a la proteína fluorescente más cercana conocida, que es una versión modificada por humanos de una proteína que se encuentra en las anémonas de mar de la punta de burbujas (Enjo cuádruple) – Criaturas marinas coloridas que parecen tener burbujas en los extremos de sus tentáculos. El resto de la secuencia es único y requeriría un total de 96 mutaciones genéticas diferentes para evolucionar. Estos cambios habrían llevado más de 500 millones de años evolucionar naturalmente, según el estudio.
Investigadores de una empresa llamaron Escala evolutiva ESMGFP presentado y el modelo AI utilizado para crearlo, ESM3, en un estudio de preimpresión el año pasado. Los científicos independientes ahora han revisado esos hallazgos, que se publicaron el 16 de enero en la revista Ciencia.
ESM3 no diseña proteínas dentro de las limitaciones habituales de la evolución. En cambio, es un solucionador de problemas que llena los vacíos de código de proteína incompleto proporcionado por los investigadores, y al hacerlo diseña algo que podría existir en función de todas las vías potenciales que la evolución podría tomar.
«Hemos encontrado que ESM3 aprende biología fundamental y puede generar proteínas funcionales fuera del espacio explorada por la evolución», coautor del estudio Alex rivesEl cofundador y jefe científico de EvolutionaryScale, dijo Live Science en un correo electrónico.
El nuevo estudio se basa en la investigación que rive y sus colegas comenzó en MetaLa empresa matriz de Facebook e Instagram, antes de comenzar EvolutionaryScale en 2024. ESM3 es su última versión de un modelo de lenguaje generativo similar al GPT-4 de OpenAI, que ejecuta CHATGPT, pero se basa en la biología.
Las proteínas están formadas por cadenas de moléculas llamadas aminoácidos, cuya secuencia es proporcionada por genes. Las diferentes proteínas tienen diferentes secuencias de aminoácidos. También difieren estructuralmente, cada uno doblando en una forma única que les permite llevar a cabo su función, según Educación en la naturaleza. Para que ESM3 comprenda las proteínas, los investigadores alimentaron los datos del modelo sobre las propiedades principales de una proteína (secuencia de aminoácidos, estructura y función) como una serie de letras.
El equipo entrenó a ESM3 en datos de 2.78 mil millones de proteínas que se encuentran en la naturaleza. Luego, los investigadores ocultaron al azar partes de un plan de proteínas y tuvieron que ESM3 enchufó los huecos para completar el código en función de lo que había aprendido.
«De la misma manera que una persona puede completar los espacios en blanco en el soliloquio» para _ o no para _, ese es el _ «, podemos entrenar un modelo de lenguaje para llenar los espacios en blanco en proteínas», dijo Rives. «Nuestra investigación ha demostrado que al resolver esta simple tarea, la información sobre la estructura profunda de la biología de las proteínas surge en la red».
Los científicos ya modifican proteínas naturales e diseñan nuevas para una variedad de fines. Por ejemplo, las proteínas fluorescentes verdes se usan ampliamente en los laboratorios de investigación. Su código genético a menudo se agrega a los extremos de otras secuencias de ADN para convertir las proteínas que codifican verde. Esto permite a los científicos rastrear fácilmente las proteínas y los procesos celulares. Rives señaló que las capacidades de ESM3 pueden acelerar una amplia gama de aplicaciones para ingeniería de proteínas, incluso con la ayuda a diseñar nuevos medicamentos.
Tiffany TaylorUn biólogo evolutivo de la Universidad de Bath en el Reino Unido que no participó en la investigación, informó sobre la versión de preimpresión del estudio para la ciencia en vivo en 2024. En su análisis, Taylor escribió que los modelos de IA como ESM3 permitirán innovaciones en ingeniería de proteínas. Esa evolución no puede. Sin embargo, también señaló que la afirmación de los investigadores de simular 500 millones de años de evolución se centra solo en proteínas individuales y no explica las muchas etapas de la selección natural que finalmente crean vida.
«La ingeniería de proteínas impulsada por la IA es intrigante, pero no puedo evitar sentir que podríamos estar demasiado seguros de asumir que podemos burlar los intrincados procesos perfeccionados por millones de años de selección natural», dijo Taylor.